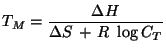>
 ') ;?>
') ;?>
 ') ;?>
') ;?>
 '); ?>
'); ?>
Next: Thermodynamisches Alignment
Up: Auswahl spezifischer Proben für
Previous: Das Probe Design Problem
Die Stabilität einer Bindung zwischen zwei DNA- bzw. RNA-Stücken
kann im Hinblick auf das Chip-Experiment am besten durch die
Temperatur ausgedrückt werden, bei der sich die beiden Teilstücke
trennen. Sie kann (mittels eines Nearest-Neighbor-Modells)
approximativ berechnet werden, unter der Annahme dass
wobei 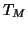 die gesuchte Temperatur,
die gesuchte Temperatur, 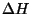 die
Enthalpie-Änderung,
die
Enthalpie-Änderung, 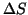 die Entropie-Änderung bei
Bindungsreaktion und
die Entropie-Änderung bei
Bindungsreaktion und 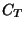 die Konzentration der DNA bzw. RNA
bezeichnet. Damit lässt sich das Probenauswahl-Problem (zunächst
für einzelne Sequenzen, unter Vernachlässigung der
Familien-Sensitivitäts-Problematik) wie folgt formalisieren: Gegeben
die Konzentration der DNA bzw. RNA
bezeichnet. Damit lässt sich das Probenauswahl-Problem (zunächst
für einzelne Sequenzen, unter Vernachlässigung der
Familien-Sensitivitäts-Problematik) wie folgt formalisieren: Gegeben
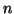 Sequenzen
Sequenzen
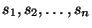 , bestimme eine Temperatur
, bestimme eine Temperatur 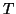 und
und
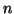 Proben
Proben
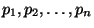 so dass
so dass
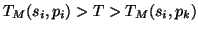
für alle
 ') ;?>
') ;?>
 ') ;?>
') ;?>
 '); ?>
'); ?>